1.1?植物組織:CTAB法
- 液氮研磨樣品,分裝至離心管中
- 向離心管中加入 CTAB,水浴
- 離心后向上清中加入氯仿/異戊醇(24:1),離心
- 向上清中加入異丙醇和乙酸鈉溶液,離心,棄上清
- 加入 75%乙醇,離心,棄上清
- 晾干,加入 TE ?溶解 DNA ,保存在-20℃冰箱
1.2?動物組織:SDS法
- 液氮研磨組織樣,分裝至離心管中
- 加入 SDS 溶液,水浴
- 加入 NaCl 溶液,靜置后離心
- 向上清中加入氯仿/異戊醇(24:1)后離心
- 向上清中加入異丙醇后離心,棄上清
- 加入 75%乙醇,離心,棄上清
- 晾干,加入 TE 溶解 DNA,保存在-20℃冰箱
1.3?動物血液:Kurabo 試劑盒法
QuickGene DNA whole blood kit S (DB-S),廠家:Kurabo,貨號:40321300101
1.4?粗體核酸純化
試劑盒名稱:QIAGEN Genomic-tip 20/G,廠家:QIAGEN,貨號:10223
試劑盒名稱:QIAGEN Genomic-tip 100/G,廠家:QIAGEN,貨號:10243
1.5?微生物樣本
使用TGuide S96磁珠法土壤/糞便基因組DNA提取試劑盒完成核酸的提取
2、檢測方法
2.1?檢測方法
- 濃度檢測
Nanodrop:賽默飛 Thermo ,型號 NANODROP2000
Qubit:廠家YEASEN, 型號CAT 12642-A ,試劑1XdsDNA HS Assay kit for Qubit
- 完整性檢測(瓊脂糖凝膠電泳)
電泳儀:廠家:天能 Tanon ,型號 EPS600
電泳槽:廠家:天根生化科技(北京)有限公司,型號:HE- 120
2.2?判定標準
- 總量≥6μg(單次建庫)
- 濃度≥100ng/ul
- 體積≥10(ul)
- OD260/280在7-2.2之間,OD260/230在1.5-3.0之間
- AGE:size≥23K,降解條帶>5K,點樣孔無或有輕微污染,N/Q≤2
- 樣本狀態正常,樣本粘稠、顏色異常、有渾濁或不溶物均不合格
注:特殊物種實驗人員會根據經驗進行判斷,具體以檢測報告中的判斷結果為準
3、建庫方法
3.1?建庫流程
- 準備高質量核酸,g-TUBE 管使核酸片段化
- 使用BluePippin進行片段篩選
- 使用 NEBNext FFPE DNA Repair Mix 和 NEBNext Ultra II End Repair/dA-Tailing Module 完成核酸片段的損傷修復和末端修復加 A
- 使用無擴增條形碼擴展試劑盒添加 barcode 序列
- 使用無擴增條形碼擴展試劑盒完成測序接頭的連接
3.2?建庫試劑
- 文庫構建試劑:NEBNext FFPE DNA Repair Mix貨號?M6630L 廠家?NEB
- 文庫構建試劑:NEBNext Ultra II End Repair/dA-Tailing Module貨號E7546L ?廠家NEB
- 文庫構建試劑:無擴增條形碼擴展試劑盒1-96 ?貨號SQK-NBD114.96 廠家Nanopore
3.3?建庫設備
- 數字 3*32PCR 儀??廠家?Appliedbiosystems???儀器規格?proflex3*32
- 制冷型程控五段金屬浴??廠家?天根生化科技(北京)有限公司??儀器規格?OSE-DB-02
4、測序方法
4.1?上機操作
- 用測序芯片制備試劑盒配制 Flow cell Priming mix
- 用測序輔助擴展試劑盒配置上機文庫
- 用 PromethION ?Flow ?Cells 芯片,在PromethION48測序儀運行MinKnow 軟件,開始測序,默認運行72 小時
4.2?測序試劑
- 測序芯片制備試劑盒??貨號?EXP-FLP004 ??廠家?Nanopore
- 測序輔助擴展試劑盒??貨號?EXP-AUX003 ?廠家?Naonopore
- 測序芯片??PromethION Flow Cell(R10.4.1)??貨號?FLO-PRO114M ??廠家?Nanopore
4.3?測序設備
PromethION48 測序儀??廠家?Nanopore???儀器規格?PromethION48
]]>ONT測序技術在多個方面具有非常強悍的優勢,然而,一份合格的下機數據才是科研成功研究的基礎,為保證得到準確的轉錄組結構分析和定量結果,需要對測序數據進行嚴格的質控評估。那么我們今天一起學習一下《Summary statistics and QC tutorial》,ONT官方提供的對測序raw?data進行全面數據質控的教程。
介紹
此教程適用于指導對單個nanopore測序芯片產出的數據進行評估,評估的主要內容如下所示:
1、測序產出(測序得到多少reads,多大數據量);
2、測序數據的質量和長度分布;
3、如果加入了barcode序列進行混樣建庫,測序數據在不同樣品的分布。
準備
直接到教程的github頁面下載或通過git命令下載:
git clone https://github.com/nanoporetech/ont_tutorial_basicqc.git QCTutorial
后續分析會用到下載目錄QCTutorial下的以下內容:
1) Nanopore_SumStatQC_Tutorial.Rmd:Rmarkdown文件,說明文檔和用于執行分析。
2) RawData/lambda_sequencing_summary.txt.bz2:示例文件,Guppy對測序reads進行堿基識別生成的相關信息文件。
3) RawData/lambda_barcoding_summary.txt.bz2:示例文件,用于區分混樣建庫時多樣品的barcode信息。
4) environment.yaml:指定分析所需軟件包及計算環境的文本文檔。
5) config.yaml:配置文件,用于指定分析所需的輸入。
2、創建Conda環境
為了方便執行分析所需軟件包及其依賴的安裝及管理,需要安裝Conda并創建用于此分析的環境。
1)?Conda安裝(Python3版本的Miniconda):
wget https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh
bash Miniconda3-latest-Linux-x86_64.sh
bash
2)?創建Conda環境及環境激活(第1步中下載的environmen.yaml用于環境初始化):
創建環境:conda env create –name BasicQC –file environment.yaml
激活環境:source activate BasicQC
分析
進行分析之前需先準備配置文件,通過修改準備步驟下載的config.yaml中相應的參數來完成,需要修改的內容主要有:
| 修改內容 | 內容說明 | 示例 |
|---|---|---|
| inputFile | 堿基識別的統計信息 | sequencing_summary.txt.bz2 |
| barcodeFile | 混樣建庫的barcode信息 | barcoding_summary.txt.bz2 |
| basecaller | 堿基識別工具 | Guppy 2.1.3 |
| flowcellId | 測序芯片ID | FAK41706 |
注:如為單樣品測序無barcode信息,則barcodeFile部分為空。
準備完成后,可以通過命令行啟動分析,命令如下:
R –slave -e ‘rmarkdown::render(“Nanopore_SumStatQC_Tutorial.Rmd”, “html_document”)’
如果習慣圖形界面操作,也可以通過Rstudio載入Rmarkdown文件執行分析:
結果
上述分析完成后會將分析結果存放至HTML文件,可用瀏覽器打開Nanopore_SumStatQC_Tutorial.html進行查看。對單個芯片約1M reads分析的部分結果展示如下(結果來自教程,堿基識別使用Guppy 2.1.3,根據識別序列的平均質量值將其分為pass和fail兩種,質量值閾值默認為7):
1、總結
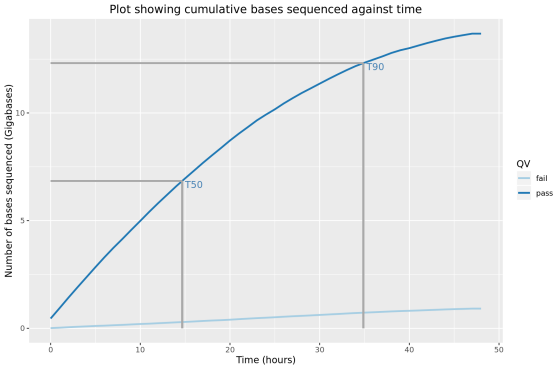
展示了數據產出的總體情況(如下圖,本分析中堿基識別共產出991,715條序列,14.6G堿基)。

2、質量長度
此部分展示了對識別出的所有序列質量和長度信息的統計結果,包括序列的平均長度,N50和平均質量,序列長度和質量的密度分布等

3、測序表現
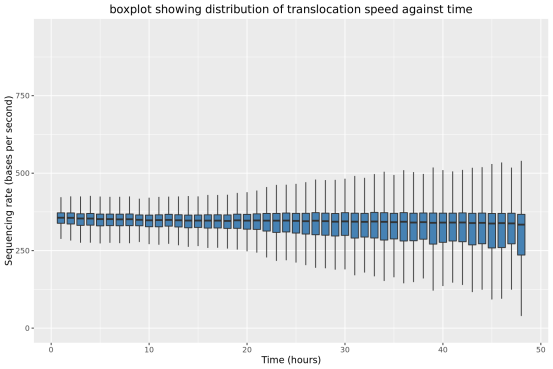
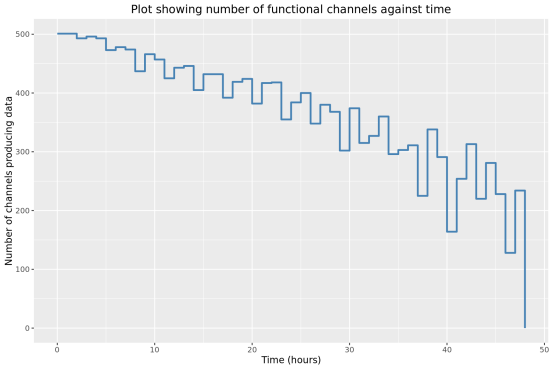
此部分內容統計了隨測序時間變化,測序累計序列個數,堿基個數,測序速度和有效工作納米孔數等指標的變化情況。

4、區分混樣
在加入barcode序列混樣測序的情況下,barcode識別區分的結果展示如下,包括barcode識別效率,區分的文庫個數及每個文庫中序列個數占比和長度信息等。

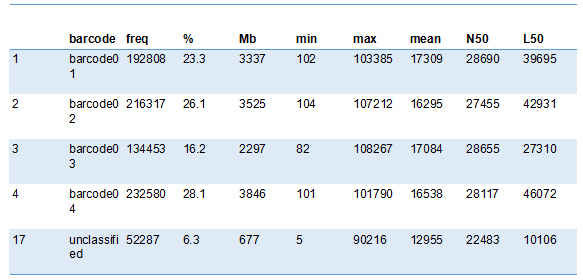
上面展示了分析結果的部分內容,更多細節的內容可參考底部的相關鏈接。
rawdata的質控評估只是整個信息分析的開始,是為了對測序數據有大致的整體認識,以便更好地指導后續分析。然而分析的每個環節都會對最終結果產生影響,因此每一步的處理都要深思熟慮。
小編寄語
2018年8月牛津納米孔公司與百邁客公司達成長期合作,擁有MinION、GridION X5和PromethION三種型號全套納米孔測序儀。至今已積累了豐富的項目經驗,全長轉錄組成功案例先后發表在《Plant Biotechnol J》、《J Hazard Mater》、《Biotechnol Biofuels》、《Sci Rep》、《Fish & Shellfish Immunology》等國際知名期刊,已發表文章研究物種分別有楊樹、吳松草、風箏果、甘薯、野生甘薯、兔子、跳甲、花羔紅點鮭和辣椒,覆蓋領域分別為林木、哺乳動物、昆蟲、水產和作物等。
如您有任何全長轉錄組等相關問題,歡迎點擊下方按鈕,我們將竭盡全力為您答疑、設計方案和提供高分成功案例等。
參考鏈接:
https@//github.com/nanoporetech/ont_tutorial_basicqc(@換成:)
https@//community.nanoporetech.com/knowledge/bioinformatics(@換成:)
]]>
